2020年5月19日,国际基因组学领域知名期刊《Genome Research》在线发表了我校前沿交叉学科研究院生物学研究中心夏庆友教授团队的最新研究成果“Genome-wide CRISPR screening reveals genes essential for cell viability and resistance to abiotic and biotic stresses in Bombyx mori”。该研究首次构建了家蚕全基因组编辑筛选平台,并且运用该平台在全基因组水平筛选鉴定了家蚕的必需基因、响应生物胁迫或非生物胁迫的基因。

我校团队于2004年完成了家蚕基因组图谱解析,使家蚕成为较早完成基因组测序的物种之一,随后又相继建立了蛋白质组、代谢组、RNAi、转基因、基因编辑等一系列家蚕功能基因组研究平台。但是,随着家蚕模式生物作用的不断加强和蚕桑丝绸产业模式的快速升级,传统的功能基因研究手段和通量已经无法满足,迫切需要新的研究手段。
该研究构建了覆盖家蚕几乎全部基因的CRISPR载体文库和家蚕细胞文库,并进行了多项突变筛选。这种基于全基因组的突变文库构建和筛选研究,对于功能基因组学和动植物品种改良都具有重要价值。作者首先构建了基于piggyBac转座子系统的家蚕细胞转基因敲除骨架载体pB-CRISPR,以此为基础,构建家蚕全基因组编辑质粒文库pB-CRISPR library,总计构建了94,000个 sgRNAs, 可以覆盖家蚕绝大多数蛋白质编码基因(16,571个基因)。作者将质粒文库稳定整合到家蚕胚胎细胞BmE的基因组上,构建成家蚕BmE细胞全基因组敲除细胞文库BmEGCKLib,该细胞文库的覆盖度超过1000倍,可以覆盖约97%的家蚕蛋白质编码基因。随后,作者运用家蚕BmE细胞全基因组敲除细胞文库执行了一系列的家蚕BmE细胞功能基因筛选,鉴定了1,006个必需基因、838个生长抑制基因、3,013个响应极端温度的基因、1,614个参与BmNPV侵染的基因,并对这些关键基因进行细致的分析和功能验证。
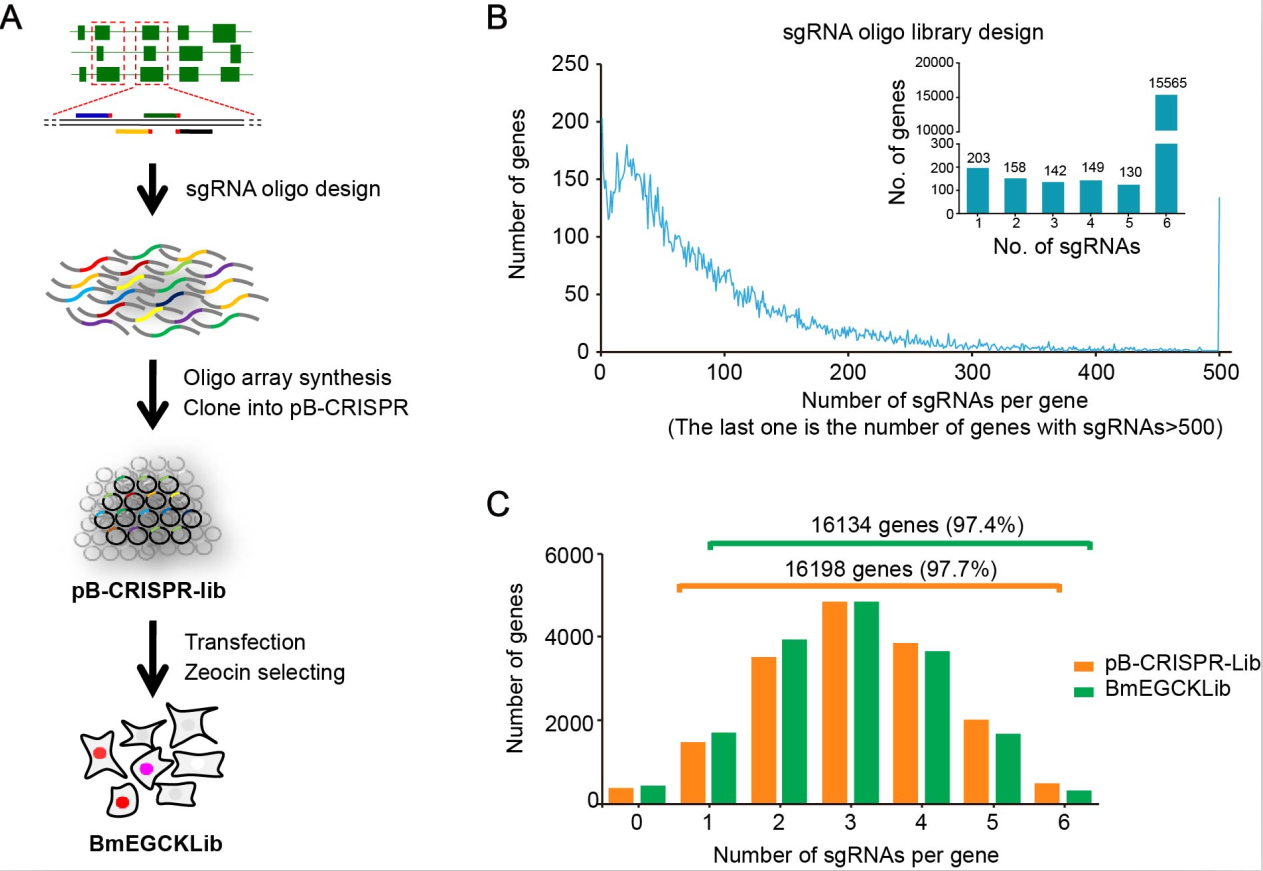
该研究为家蚕功能基因研究提供了一种全新的、不设前提、靶标导向的研究方法,将极大的推动家蚕功能基因研究进入规模化、自主知识产权化的新阶段,促成家蚕遗传育种的精准化、规模化、工程化和多元化,为产业创新升级提供新的思路和素材。此外,piggyBac转座子系统的宿主范围很广、转座效率很高,因此该研究也为其他物种构建高通量筛选平台提供了重要借鉴。
博士研究生常珈菘和王若琳为论文的共同第一作者,通讯作者为马三垣副教授和夏庆友教授。本研究得到了国家自然科学基金、双一流学科建设经费和重庆市科技局项目的支持。
论文链接:https://genome.cshlp.org/content/early/2020/05/18/gr.249045.119.abstract